编者按:
为推动我国以IPv6为核心的下一代互联网技术的创新与普及,在教育部科技发展中心的指导下,自2015年起,赛尔网络有限公司每年出资2000万元,面向全国高校举办“下一代互联网创新项目”和“下一代互联网技术创新大赛”,鼓励扶持以大学生为核心的下一代互联网技术创新,以推动中国IPv6网络技术的普及与发展。
自此活动开展三年以来,到2017年底,创新项目完成了三期项目遴选以及首批项目验收,共支持了四百多个项目,总资助额达4000多万;三界创新大赛共有来自全国100余所高校的500余个项目参加角逐,评选出了众多优秀的下一代互联网创新项目。
为了更好地总结该项目所取得的成果经验,同时呼应两办发布的《下一代互联网规模部署计划》,进一步做好下一代互联网技术的宣传与推广工作,自本期起设立“IPv6创新”专栏,以发掘该项目中的亮点方案与明星团队,回顾总结该项目取得的经验与成果,发掘下一代互联网创新发展可资借鉴的规律与趋势,为促进下一代互联网技术创新、成果转化与人才培养做好指引方向。
在飞速发展的网络信息时代,大数据已经渗透到医学、药学、化学、生物和物理等各个学科,很多创新的先进信息技术不断促进这些领域的快速发展。为了认真贯彻和落实这项政策并推动下一代互联网在高校的全面发展,在大学课堂上普及并加强IPv6知识和各学科的深度融合显得尤为重要。基于IPv6的教学媒介是IPv6教育的基础,但是目前高效灵活的IPv6网络教学平台并不多见。
C6C IPv6IaaS云服务平台
C6C IPv6IaaS云服务平台是赛尔网络打造的新一代服务器租用产品,它基于IaaS的云服务架构,具有可弹性伸缩、管理高效、稳定安全和低IT 综合成本等特点。该平台可以通过网页、Xshell或VNCViewer进行连接,这种方式便于在课堂上展示IPv6的相关知识,让学生更深入地了解基于IPv6的各种应用。具体的申请过程如下:
首先,登录赛尔网络的云服务器(http://yun.n.edu.cn)。可以根据教学需要申请基于Linux和Windows操作系统的云服务器。
其次,使用申请的账户和密码通过浏览器登录到C6C IPv6IaaS云服务器主机的管理界面。通过管理界面可以看到申请到的主机名和IPv6地址等信息。Linux操作系统的服务器管理界面显示IPv6地址有8组,每组有3或4个16进制的数字;而Windows系统显示了IPv6地址的一种缩写形式,缩写地址2001:da8:270:2019::52对应的完整地址为:2001:0da8:0270:2019:0000:0000:0000:0052。通过该界面可以让学生了解IPv6地址的一些特征和缩写方式。
最后,登录C6C IPv6IaaS的云服务器主机。通过主机链接下面的“远程链接”就可以登录到对应的服务器上。若是想刷新缓存或者出现死机现象,可以通过点击“重启主机”的链接进行操作云端服务器。
基于IPv6网络的药物虚拟筛选教学平台
药物的虚拟筛选可以通过利用IPv6网络上的空闲电脑进行计算,如果在课堂上讲述整个流程,往往让学生感觉抽象并难以理解其内容。一般课堂时间是45分钟左右,微课堂只有5~8分钟,这就需要用一个高效的教学平台,C6C IPv6IaaS云服务器提供了真实的IPv6网络环境,在此基础上,能快速搭建基于IPv6网络的药物虚拟筛选平台,从而可以快捷地在课堂上演示整个药物虚拟筛选的过程;同时,由于C6C IPv6IaaS云服务器的操作便捷性,可以在课堂上让学生亲自体验整个药物虚拟筛选的流程,这样不仅会给学生留下深刻的印象,而且也有助于老师提高讲课效率。
基于IPv6的药物虚拟筛选教学平台需要药物分子、蛋白靶点、IPv6的网络环境和相关软件等。药物分子一般选自ZINC 数据库(http://zinc.docking.org),蛋白靶点一般选自PDB数据库(http://www.rcsb.org);C6C IPv6IaaS平台提供IPv6网络环境;使用Apache FtpServer、MolGridCal和JPPF分别在IaaS云服务器上搭建数据传输中心和部署药物的虚拟筛选。
IPv6网络下的药物虚拟筛选具体实现流程如图1所示。首先,设计药物虚拟筛选程序MolGridCal(https://github.com/MolGridCal/MolGridCal),MoGridCal 是基于JPPF 并行计算框架开发的程序,可以在IPv6网络上部署药物的虚拟筛选。它的基本原理是通过发送Autodock Vina的药物虚拟筛选任务到服务器(Servers)端,控制Servers端分配任务到不同的计算节点(nodes)上,而计算节点根据Servers端传输过来的命令从FTP服务器上下载药物分子进行Autodock Vina的药物虚拟筛选,筛选好的结果再上传到FTP指定的目录中,这样就完成了整个药物大数据的虚拟筛选;除此之外,MolGridCal还可以通过设计算法进行分配药物虚拟筛选的任务,这一点可以通过软件Notepad++在课堂上进行修改并展示。
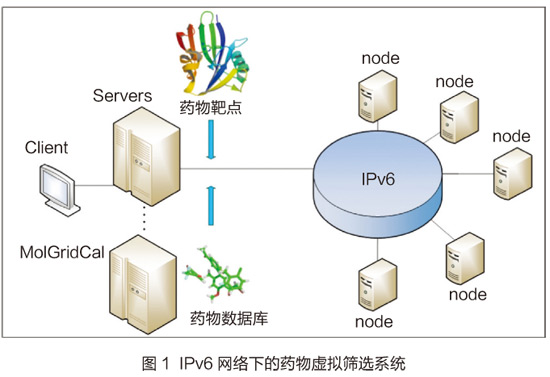
其次,配置计算节点(nodes)。在nodes的配置文件里,添加连接Servers端的IPv6地址,如果在有安全防护的内网里面,可以选择11111端口进行连接,为了提高安全级别,也可以选择使用SSL安全证书在11443端口进行连接;同时在nodes端可以选择利用空闲资源的模式,即当敲击鼠标和键盘时,此时nodes端会认为系统处于繁忙状态,停止药物虚拟筛选的任务,当nodes端没有任何操作,到达设定的空闲时间阈值,nodes端又会重新启动停止的任务,这样就可以达到充分利用电脑空闲资源的目的。此外,nodes端存放了蛋白质受体靶点和Autodock Vina程序等,当Server端传来任务指令时,计算节点启动Java虚拟机建立一个线程,线程启动Autodock Vina进行药物的虚拟筛选,完成的筛选结果根据程序指令再传输到FtpServer端指定的目录中。
最后,配置Servers端。在Servers端运行程序监听11111和11443端口,11111端口用于监听一般计算节点的连接,11443端口用于监听SSL安全证书的计算节点连接,Servers端既可以接收来自MolGridCal的任务,也可以接收到nodes端的连接消息,不同的Servers端可以相互连接互不影响。另外在Servers端提供了manual、autotuned、proportional和nodethreads四种分配任务的算法。如果把大量的虚拟筛选任务同时分配到nodes端,会占据nodes端的大量内存,为了尽可能减轻nodes端的负担,选择manual算法进行药物虚拟筛选的任务分配,每次只分配一个任务到单个node端中,计算完毕后再进行分配,这样就可以根据nodes端的计算能力动态分配虚拟筛选任务。
由于C6C IPv6IaaS云服务平台可以提供固定的Servers地址和相对稳定的网络环境,所以组建的药物虚拟筛选系统不仅稳定而且具有很好的教学示范效果。
交叉学科教学演示
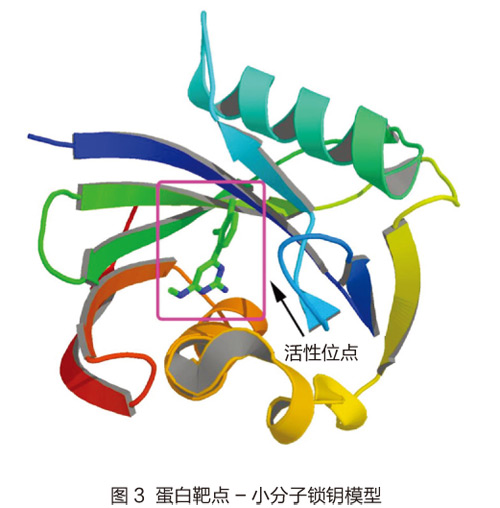
药物虚拟筛选的教学平台涉及到IPv6网络、医药、生命科学、物理和化学等多个学科。要想把这些内容全部在大学课堂中形象地展示出来,需要很完善的演示平台。据测试,将药物虚拟筛选的程序组件部署到C6C IPv6IaaS云服务平台上,如图2所示,只需要四个步骤就可以形象地演示整个药物虚拟筛选的流程:启动FtpServer、运行Servers端进行监听、nodes端进行连接和MolGridCal进行药物的虚拟筛选;通过Web网页打开IaaS云服务器到最终的药物虚拟筛选演示可以控制在8分钟以内,这样不仅可以形象地给学生展示基于IPv6网络的药物虚拟筛选流程,而且大大提高了大学课堂教学的效率。FtpServer端、Servers端、nodes端、MolGridCal都是使用Java程序完成的,有良好的跨平台性质,任何一个程序组件都可以部署到Windows或Linux的系统平台上,这样就可以在大学课堂上给学生讲解软件跨平台的相关知识和应用;除此之外,本系统的药物虚拟筛选是基于分子对接的技术,分子对接的基本原理是锁-钥模型,如图3所示。蛋白质相当于锁,药物分子相当于钥匙,形象地说,能把“锁”打开的“钥匙”就是结合效果较好的药物分子;同时分子对接技术涉及到打分函数和构象搜索算法,打分函数涉及到物理化学和数学拟合方面的知识,构象搜索包含信息学中的求解旅行商(TSP)问题和化学方面的知识;所以,结合部署在C6C IPv6IaaS云服务平台上的药物虚拟筛选系统,可以在课堂上高效、形象地讲述IPv6、最优算法、医药、物理和化学等相关知识。
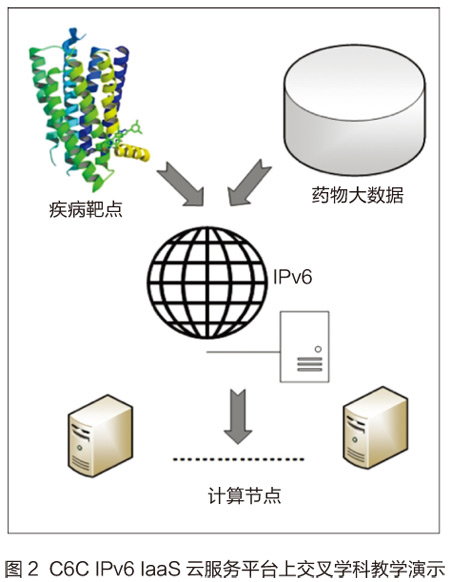
在国家大力推动IPv6部署的背景下,在具体的教学实践中我们发现C6C IPv6IaaS云服务平台不仅可以应用于交叉学科的教学,而且还可以高效地展示整个分布式网络的药物虚拟筛选流程,从而大大地节约课堂时间。(责编:杨燕婷)(作者单位为兰州大学)
本文刊载于《中国教育网络》2018年4月刊

特别声明:本站注明稿件来源为其他媒体的文/图等稿件均为转载稿,本站转载出于非商业性的教育和科研之目的,并不意味着赞同其观点或证实其内容的真实性。如转载稿涉及版权等问题,请作者在两周内速来电或来函联系。
IPv6应用论坛在福州成功举办2023/11/30
IPv6技术论坛在福州召开2023/11/29
第三届IPv6下一代互联网技术创新与国际标准研讨会在京成功召开2023/11/06
携手共促我国IPv6创新发展2023/10/09
IPv6动态丨AWS开始对IPv4全面收费,9名中国专家入选IPv6 Forum Fellow……2023/10/09
IPv6助力产业数字化转型2023/09/27
中国CNGI项目20年大事记2023/09/19









 京公网安备 11040202430174号
京公网安备 11040202430174号